03.11.2022
![]()
Bienenpollen als Indikatormatrix zur Katastererfassung pflanzlicher Biodiversität
Dr. Thomas Letzel , Analytisches Forschungsinstitut für Non-Target Screening GmbH (AFIN-TS)
Karl-Heinz Jansen, SYKAM Chromatographie Vertriebs GmbH
Stefan Ratering, Institut für Angewandte Mikrobiologie am iFZ der Uni Gießen
Bienen sind sehr fleißige Pollensammlerinnen. Sie erkunden auf der Suche nach Nahrung einen Umkreis von bis zu fünf Kilometern, und jede sammelt an schönen Tagen in ihren Pollenkörbchen bis zu einem halben Gramm Blütenstaub bzw. Pollen. Dabei werden von den Sammlerinnen blühende Pflanzen, genauer Insektenbestäuberpflanzen, angeflogen und die Pollen im sogenannten Pollenhöschen in den heimischen Stock transportiert.
Das zum Jahr 2021 gestartete Konsortium Bee2diversity macht sich die Pollensammlung der Bienen zunutze und untersucht anhand systematisch genommener Pollenhöschen die so beprobten Pflanzen und erfasst damit die Biodiversität. Im Rahmen der Initiative werden ein professionelles automatisiertes Probenahmesystem für Pollenhöschen entwickelt, zukunftweisende analytische Untersuchungsmethoden angewandt sowie ein Proben- und Analyse-Handhabungskonzept erstellt.
Die Analysestrategien umfassen die Techniken 'Imaging durch Mikroskopie', 'Hochdurchsatzsequenzierung auf genetischer Ebene' und Pflanzenmetabolomstudien mittels 'massenspektrometrischem Non-Target Screening'. Gleichzeitig werden die Informationen in einer neuartigen Laborerfassungsstrategie auf Grundlage von QR-Codes in Blockchain-Technologie eingebunden und zum Abgleich genutzt.
Das Konsortium entwickelt, etabliert, nutzt die Lösungen dabei sehr komplementär und evaluiert die Notwendigkeit der einzelnen Anwendungen bzw. die Übertragbarkeit der unterschiedlichen Analysetechniken zur nachhaltigen und wirtschaftlichen Bestimmung von Biodiversität in der beprobten Umgebung. Im Erfolgsfall soll die Technik die lokale Erfassung von Biodiversität ermöglichen und letztlich auch Imkern einen besseren Einblick in die Vielfalt der lokalen von Bienen besuchten Trachtpflanzen geben.
Das Projekt Bee2Diversity
Die vom BMWi geförderte (und als ZIM-Kooperationsprojekt vom Projektträger der AiF Projekt GmbH unterstützte) Projektstudie Bee2Diversity entwickelt ein Verfahren mit automatisierter Probenahme und Analysentechniken, das es erlaubt, ein flächen- und zeitabhängiges Biodiversitätskataster für (blühende,) bienenbestäubte Pflanzen durch Beprobung und Analyse von bienengesammelten Pollen zu etablieren. Dabei kommen modernste Analysetechniken - zunächst getrennt - zum Einsatz. Die daraus generierten Erkenntnisse werden im Laufe des Projektes miteinander kombiniert und so versucht biodiversitätsbestimmende Faktoren zu erfassen.Dabei sollen die eingesetzten Techniken und Anwendungen folgende Aussagen ermöglichen:
- Nahrungsangebot der Bienen im Bestäubungsgebiet quantitativ erfassen (d. h. Masse/Zeit);
- Umfangreiche Probenstandortcharakterisierung zur Erfassung sogenannter 'Metadaten', wie Wetterlage, Feuchtigkeit, Sonnenstunden usw. einrichten.
- Probenflüsse und Ergebnisse werden in einer neuartigen digitalen Strategie erfasst, zusammengeführt und in Kombination miteinander genutzt.
- Die Bestimmung der Pflanzenarten wird durch mikroskopische Analyse der gesammelten Pollenkörner qualitativ durchgeführt.
- Die Diversität der Pflanzen wird mittels Sequenzierung der DNA des Cytochrom c Oxidase-Gens aus den gesammelten Pollen qualitativ erfasst.
- Die Diversität der Bakterien und der Pilze in den Pollenhöschen wird mittels 16S rRNA Gensequenz- bzw. pilzliche ITS-Sequenzanalysen qualitativ erfasst.
- Diversitätsanalysen von Pflanzen-, Bakterien-, und Pilzspezies erlauben Hinweise auf Habitatqualitäten für Bestäuberinsekten.
- Eventuelle Nachweise der Bienen-Faulbrut-Erreger Melissococcus plutonius und Microsporium Nosema ceranae auf Pollenproben werden mittels spezifischer quantitativer PCR erbracht (insbesondere aufgrund entsprechender Beobachtungen von Imkern).
- 'Metabolom-Fingerabdrücke' der Pollenextrakte werden mittels Non-Target Screening Analytik unter Nutzung der RPLC-HILIC-MS/MS Technologie identifizierend erfasst.
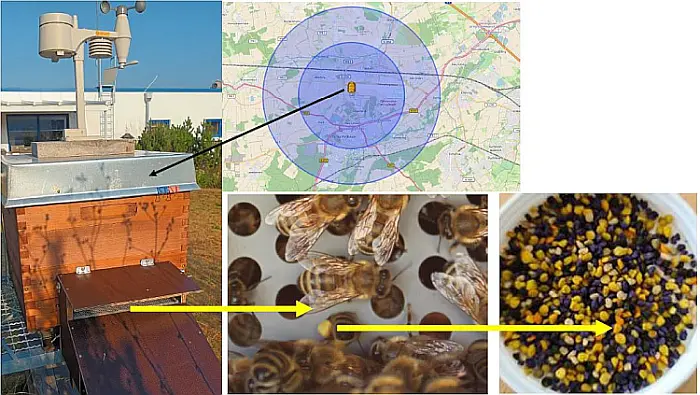
Abb.1: Links ist eine Bienenbeute mit Wetterstation zu sehen. Vor dem Flugloch ist ein Pollensammler installiert. Das mittlere Bild (unten) zeigt das Scherengitter, das für die Pollensammlung heruntergeklappt wird. Beim Hindurchkriechen streifen die Bienen die Blütenpollen ab, sodass diese aufgefangen und gesammelt werden können (rechts). Die obere Abbildung zeigt den ungefähren Flugradius der Bienen um die aufgestellte Beute im Projektgebiet.
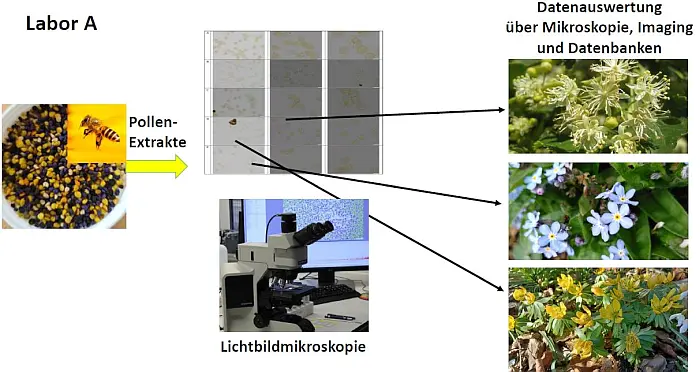
Abb.2: Von den gesammelten Pollen werden in Labor A Lichtmikroskopaufnahmen angefertigt, die im Vergleich mit Pollendatenbanken Rückschlüsse auf die beprobte Flora zulassen.
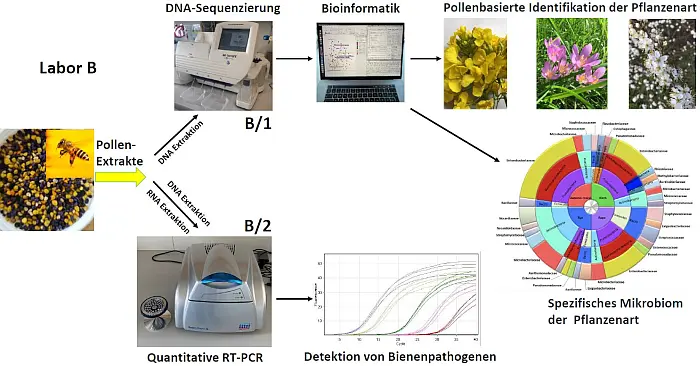
Abb.3: Von den gesammelten Pollen werden die Extrakte in Labor B mittels DNA-Sequenzierung (B/1) und quantitativer real-time PCR (B/2) analysiert, die im Vergleich mit Datenbanken Rückschlüsse auf die beprobte Flora und das Pollenmikrobiome und auf Bienenpathogene zulassen.
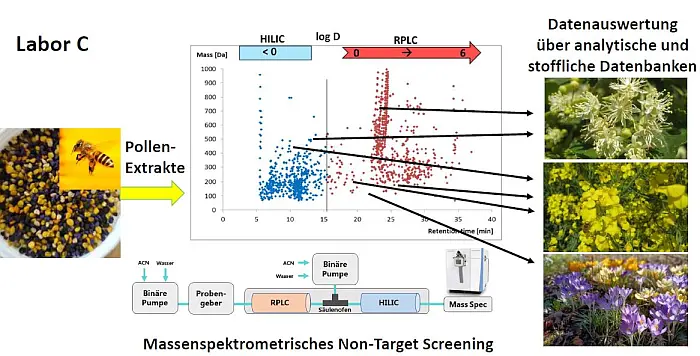
Abb.4: Von den gesammelten Pollen werden die Extrakte in Labor C mittels polaritätserweiterten Non-Target Screening mit RPLC-HILIC-HRMS/MS auf enthaltene Moleküle untersucht, die im Vergleich mit analytischen und stofflichen Datenbanken Rückschlüsse auf die beprobte Flora zulassen.
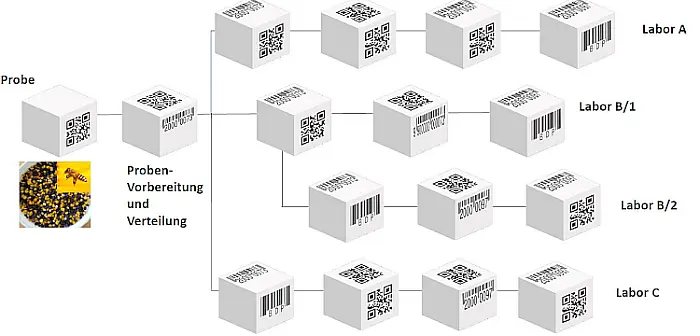
Abb.5: Schematische Skizze des Proben- und Labormanagementsystems mit QR-Codes und unter Nutzung der BlockChain Methode (für alle drei eingebundenen Labore).
Erste Ergebnisse
Die Ergebnisse in der ersten Projekthälfte der Projektstudie zeigen erfolg- und informationsreiche Erkenntnisse und stellen sich wie folgt dar:
Es gibt nun ein Verfahren mit Probenahme und Analysentechnik, das es erlaubt, ein flächen- und zeitabhängiges Biodiversitäts-Kataster für (blühende) Pflanzen zu etablieren. Der Einbau von ersten kommerziellen Pollensammlern an ausgewählten Bienenstöcken ist erfolgt und liefert eine reproduzierbare Sammlung von Probenmaterial. Die Probenvorbereitung von Pollen zur Nutzbarkeit für die analytischen Methoden der drei Labore wurde untersucht, angepasst und harmonisiert so dass im folgenden Projektzeitraum daran festgehalten werden kann.
Mikroskopische Untersuchung von Pollen in den ersten beiden 'Sammelperioden' führen zum Aufbau einer ersten bildgestützten Datenbank für standardisierte Erkennung.
Die Entwicklung einer standardisierten Methode zur Erfassung der Pollendiversität und des Pollenmikrobioms inklusive Krankheitserreger für Bienen (mittels Hochdurchsatzsequenzierung von spezifischen Amplikons aus der Pollen-DNA und mikrobieller DNA und qPCR) wurde gestartet und erfolgreich etabliert. Dabei wurde in den ersten zwei Sammelperioden an zwei Standorten die Diversität der Pflanzen und der mikrobiellen Besiedlung der gesammelten Pollen bestimmt und die Diversitätsanalyse der Pflanzen mit der klassischen mikroskopischen Methode zur Pollenidentifizierung verifiziert.
Die Weiterentwicklung einer polaritätserweiterten Non-Taget Screening (NTS) Analyse mittels RPLC-HILIC-MS/MS für Pollenextrakte wurde durchgeführt und ist abgeschlossen, so dass diese im weiteren Projektzeitraum Anwendung findet. Ebenso wurden Messstrategien implementiert, die zukünftig einen erhöhten Probendurchsatz erlauben. Auswertestrategien (zur potentiellen Identifizierung und statistischen Nutzung speziesspezifischer Moleküle der unterschiedlichen Pflanzenmetabolome) wurden konzipiert und zur weiteren Nutzung etabliert. So ist die Zuordnung dieser 'Pollenmetabolom'-Ergebnisse zu Pflanzenspezies grundsätzlich möglich und schließlich zur späteren Katastererfassung nutzbar. Die Ergebnisse der ersten beiden Sammelperioden liefern nun Erkenntnisse, die im dritten Jahr Eingang finden.
Der Aufbau einer Plattform zum digitalen Abgleich sämtlicher erfasster Daten, Ergebnisse sowie Erkenntnisse wurde konzipiert und gestartet. Diese Plattform bildet die Basis zur Erstellung eines Pflanzenkatasters und die Charakterisierung der pflanzlichen Biodiversität in den erfassten Regionen.
Die Bildung eines Netzwerks mit Imkern, Forschungseinrichtungen und Verbänden wurde gestartet und soll auch zukünftig aktiv forciert werden. Dabei werden diese über die Projektlaufzeit (und darüber hinaus) die Studie mit ihren Bienenvölkern unterstützen und ihre Beobachtungen zur Bienengesundheit (Faulbrut und Nosema: Koloniebefall sowie Mortalität) erfassen und dem Konsortium mitteilen.
Zusammenfassung
Durch Bienen gesammelte Pollen sollen als Indikatormatrix zur Katastererfassung pflanzlicher Biodiversität herangezogen werden. Die Untersuchung der Pollen unterschiedlicher Pflanzen mit modernen Analysetechniken eröffnet einen detaillierten Blick auf die Biodiversität der Flora in dem definierten Radius. Schwerpunkte liegen hierbei während der gesamten Laufzeit auf automatischer Pollensammlung, Analysetechniken wie Mikroskopie, genetischer Molekularanalytik und massenspektrometrischem Non-Target-Screening. Alles zusammen wird verknüpft durch moderne Datenerfassung über QR-Codes und deren Nutzung in Blockchain-Technologie.
Die bisherigen Erkenntnisse sollen im zweiten Projektabschnitt verstärkt zur professionellen Erfassung genutzt und etabliert werden, so dass die Katastererfassung als Idee auch nachhaltig in der Realität umgesetzt werden kann. Wir freuen uns über Kontaktaufnahme und Diskussionen.
Danksagung
Das FuE-Kooperationsprojekt (mit dem Förderkennzeichen KK5035801SA) wird über die AiF Projekt GmbH im Rahmen des Programms "Zentrales Innovationsprogramm Mittelstand" (ZIM) vom Bundesministerium für Wirtschaft und Energie aufgrund eines Beschlusses des Deutschen Bundestages gefördert.
